作科所搭建大豆种质资源组学数据库SoyFGB v2.0
近日,中国农业科学院作物科学研究所的水稻分子设计技术与应用创新团队和大豆优异基因资源发掘与创新利用团队联手,升级功能基因组育种FGB(Functional Genomics Breeding)范式,联合搭建大豆代表性种质组学数据库,为将我国大豆种质资源优势转变为基因资源优势和品种优势提供重要信息平台和挖掘工具。8月2日,相关研究成果在线发表在《Science Bulletin》。
据邱丽娟研究员介绍,种质资源是作物育种的重要物质基础,是国家战略性资源。大豆起源于中国,我国大豆种质资源在世界上最为丰富。在这个组学时代,海量多组学数据不断产生,如何充分共享组学数据是一直困扰着大豆种质资源工作者的重要问题。
研究团队在2214份代表性大豆种质资源(2K-SG)重测序(SCIENCE CHINA Life Sciences, 2022)和表型采集基础上,采用自主创立的FGB范式开展数据平台搭建工作。该范式于2015年发布在《科学通报》,首次应用于水稻数据库构建(https://www.rmbreeding.cn),并于2020年完成首度升级(Plant Biotechnology Journal,2020)。针对大豆在基因组和种质资源等方面的特点及用户需求差异,研究团队再次升级并拓展FGB范式,建立了SoyFGB v2.0数据平台(https://sfgb.rmbreeding.cn)。
SoyFGB v2.0数据平台(https://sfgb.rmbreeding.cn)
SoyFGB v2.0数据平台的先进性主要表现在以下3个方面。一是提供离散值的表型数据来帮助用户识别用于育种或遗传研究的“有用”种质资源,实现了2K-SG的33个数量性状与9个质量性状的非下载共享。二是用户可以利用SoyFGB或用户自有的未公开表型数据来实现表型和单倍型变异的相关性在不同基因组分辨率下的在线解析。三是一旦获得基因组作图定位与表型性状相关区域,使用“搜索”和“浏览”模块,用户可以获取2K-SG 的基因组变异,用于实验验证。根据用户实际体验,与传统Excel表格辅助进行单倍型分析相比,采用SoyFGBv2.0进行特定基因的单倍型分析能够提高效率近60倍。总之,SoyFGB v2.0的发布将不仅极大地促进大豆种质资源的单倍型挖掘,而且为大豆种质资源组学数据共享和挖掘提供一个通用新范式。
作科所郑天清副研究员和李英慧研究员为论文共同第一作者,邱丽娟研究员为通讯作者。该研究得到国家重点研发计划、国家自然科学基金、中国农业科学院国际合作、创新工程、基本科研业务费(平台项目)和比尔盖茨基金项目资助。
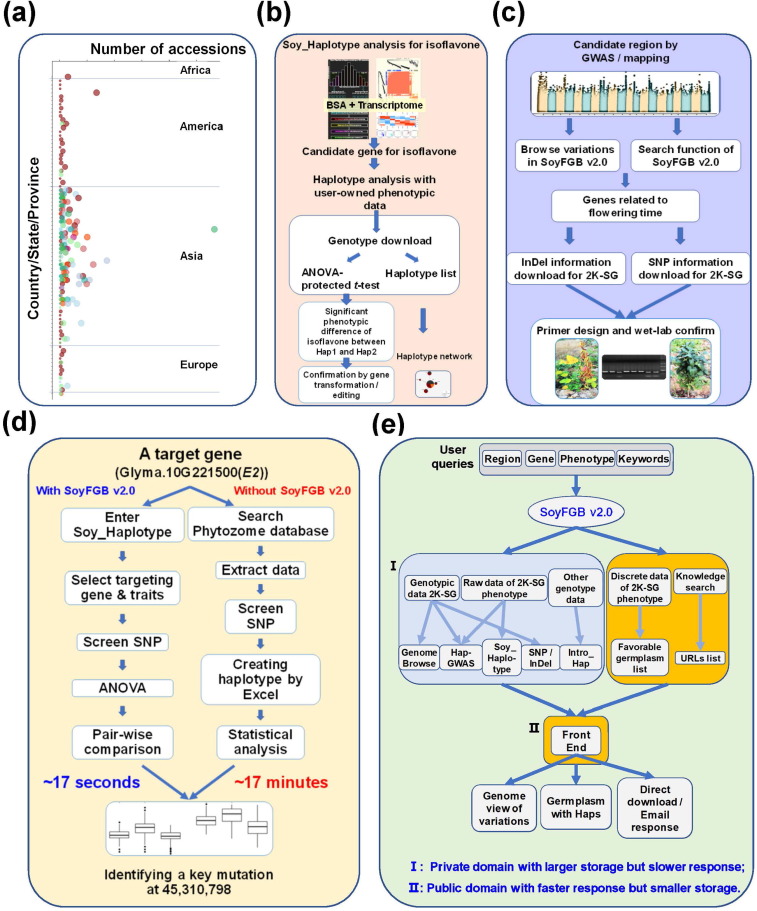
用户案例以及SoyFGB v2.0 的结构




